プレスリリース
明治大学 越水静助教、基礎生物学研究所 南野尚紀特任助教、金沢大学 西山智明助教、立教大学 養老瑛美子助教、理化学研究所 佐藤繭子技師ら共同研究チームはこのたび、オミクス解析によるスクリーニングから、ゼニゴケにおいて精子形成に関与する新規因子BLD10を発見しました。さらに、BLD10遺伝子の精子形成における機能は、BLD10遺伝子が分子進化した結果、祖先機能に加えて新たに獲得した機能である可能性を示しました。
本研究成果は、英国の国際雑誌「New Phytologist」2022年8月3日付(日本時間8月4日)に掲載されました(オンライン版が2022年7月16日に先行公開されました)。
本研究では、オミクスデータを活用し、それらを統合解析することで、効果的に目的遺伝子をスクリーニングしました。機能解析については、遺伝子導入系が確立されており、精子を形成するゼニゴケとヒメツリガネゴケを使用すること、および高い観察技術によって可能となりました。特に連続切片自動撮像システムを搭載した電子顕微鏡を用いて連続切片観察を行うことで、基底小体周辺の構造の詳細な解析に成功しました。
■ 本研究のポイント
・公開されているオミクスデータや複数のインシリコ解析を組み合わせることにより、精子の形成に関与する候補因子BLD10を選抜した。
・コケ植物の中でも特に様々な実験手法の適用が可能なゼニゴケとヒメツリガネゴケにおいてBLD10遺伝子の機能を解析した結果、BLD10遺伝子は鞭毛の形成起点である基底小体の構造維持に機能し、BLD10遺伝子の変異体(bld10変異体)では、基底小体の崩壊と、鞭毛の形成不全を引き起こした。
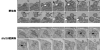
・ゼニゴケのbld10変異体は、通常、精子形成中に起きる核の凝集および伸長と、細胞質の消失に異常を示し、精子形成に著しい欠陥が見られた(図1B)。
・ゼニゴケやヒメツリガネゴケのBLD10遺伝子は、動物やクラミドモナスで基底小体の形成に機能するBLD10/CEP135ファミリー遺伝子のオーソログである。
・陸上植物のBLD10遺伝子は、陸上植物に近縁な藻類よりも進化速度が約2倍に上昇していることから、ゼニゴケの精子形成時における機能は、急速な分子進化の結果新規に獲得した可能性がある。
画像1: https://www.atpress.ne.jp/releases/321017/img_321017_1.png
図1. ゼニゴケ精子の表現型解析
A, ゼニゴケ精子の模式図。
B, 野生株(左)およびbld 10変異体(中央、右)の精子。
青:核(Hoechst33342); スケールバー:10 μm。
■ 研究の背景
卵生殖は、雌性配偶子(卵)と雄性配偶子(精子)を利用した有性生殖の一様式です。卵生殖は動物、菌類、植物にて独立に進化しており、精子の形成を含む卵生殖がどのような因子の進化によってそれぞれの系統で獲得されたのかは解決するべき大きな問いです。
ストレプト植物門(陸上植物と陸上植物に近縁の藻類を含む分類群)では、シャジクモ類、コレオケーテ類、コケ植物、小葉類、シダ類、一部の裸子植物が有性生殖に精子を用います。これらの植物の精子は、凝縮し細長く伸びた螺旋状の核(裸子植物は除く)、帯状に微小管が配向するスプライン、多層構造体(MLS; 最上層がスプラインの連続体)、複数の鞭毛など、特徴的な構造を有しています(図1A)。これまでに構造的特徴はよく研究されているものの、植物の精子形成に関わる因子の多くは未解明でした。
■ 研究内容
1. インシリコ解析による遺伝子スクリーニング
近年、DNA塩基配列解読技術の飛躍的向上により、ストレプト植物のゲノムやトランスクリプトームデータ(包括的遺伝子発現情報)が大量に蓄積されています。私たちはそれらのデータを活用し、植物の精子形成に関わる因子の候補を選抜しました。主に行った解析は、オルソグループ解析とトランスクリプトーム解析です。オルソグループ解析では、精子を作る植物にはあるが精子を作らない植物にはないオルソグループの遺伝子に焦点を当て、トランスクリプトーム解析では、精子を作る器官である造精器で特異的に発現する遺伝子に注目し、遺伝子群を選抜しました。最終的に、BLD10遺伝子を最も有力な候補遺伝子とし、更なる解析を行いました。
BLD10タンパク質は鞭毛の形成起点である基底小体のタンパク質(BLD10/CEP135)と弱い類似性を持ちますが、コケ植物における既知の情報はほとんどありませんでした。
2. コケ植物におけるBLD10遺伝子の機能解析
BLD10遺伝子の機能を調べるために、コケ植物であるゼニゴケにてBLD10遺伝子をノックアウトし、表現型解析を行いました。その結果、bld10変異体では異常な精子が形成され、通常、精子形成中に起きる核の凝集および伸長と、細胞質の消失に異常を示しました(図1B)。また鞭毛の形成にも欠陥が見られ、多くの精子が鞭毛を欠いていました。さらに連続切片自動撮像システムを搭載した電界放出型走査電子顕微鏡を用いた連続切片法にて基底小体を観察したところ、bld10変異体では開裂した基底小体などの異常な構造が観察されました(図2)。一方、ゼニゴケとは異なる分類群のコケ植物であるヒメツリガネゴケにおいてもBLD10遺伝子をノックアウトしたところ、鞭毛の形成と基底小体にのみ異常が見られました。
次に、ゼニゴケとヒメツリガネゴケにおいてBLD10タンパク質の細胞内局在を調べた結果、鞭毛の基部に位置する基底小体と密接して局在していました。
画像2: https://www.atpress.ne.jp/releases/321017/img_321017_2.png
図2. ゼニゴケ精子における連続切片電子顕微鏡像
野生株では2つの基底小体の構造が観察されるが(矢尻白)、bld10変異体では1つは構造が完全に壊れ識別できず、もう1つは基底小体が開裂していた(矢尻黒)。
スケールバー:200 nm
3. BLD10遺伝子の系統解析
前述したように、私たちが同定したBLD10タンパク質とBLD10/CEP135ファミリータンパク質の間には弱い類似性が検出されました。BLD10/CEP135ファミリータンパク質は基底小体のタンパク質であり、鞭毛を持つ藻類であるクラミドモナスでは、欠失すると基底小体が崩壊し、結果鞭毛が消失します。つまり、本研究で同定したBLD10タンパク質の鞭毛や基底小体における機能はBLD10/CEP135ファミリータンパク質と同様であることがわかりました。さらに多様な生物で相同遺伝子を探索し、遺伝子構造を推定し、翻訳したアミノ酸配列で系統解析を行った結果、ゼニゴケやヒメツリガネゴケのBLD10遺伝子は、クラミドモナスのBLD10遺伝子のオーソログであると推定されました(図3)。
さらに、陸上植物におけるBLD10遺伝子の進化速度が陸上植物に近縁な藻類(ストレプト藻類)に比べ約2倍に上昇していることがわかりました。ゼニゴケBLD10遺伝子における精子形成時の機能はゼニゴケだけに見られることから、ゼニゴケにて新規に獲得された機能であると推察され、進化速度の上昇と関連する可能性があります。
画像3: https://www.atpress.ne.jp/releases/321017/img_321017_3.png
図3. BLD10/CEP135ファミリータンパク質の系統樹
赤色下線のゼニゴケ(Marchantia polymorpha)、ヒメツリガネゴケ(Physcomitrium patens)、 クラミドモナス(Chlamydomonas reinhardtii)は機能が解析されている遺伝子。ほぼ全ての生物で一つの遺伝子で、生物の系統と大きく矛盾しないので、イチョウの二遺伝子がイチョウの系統で独自に遺伝子重複している以外、全てオーソログであると考えられる。
■ 今後の展望
ゼニゴケのBLD10遺伝子は精子形成において機能することが明らかになりましたが、その作動機序はまだ不明であり、今後の解明が待たれます。また、BLD10遺伝子は陸上植物において進化速度が上昇しており、他の陸上植物においても何らかの機能進化が起きている可能性があるため、今後の解析が期待されます。
本研究で行ったスクリーニングにより、BLD10遺伝子の他にも精子形成に関連する候補因子を選抜済みです。それら因子の機能解析によって、さらなる精子形成関連因子を同定できる可能性があります。
■ 用語解説
オミクスデータ:網羅的な生体分子についてのデータであり、具体的にはゲノムやトランスクリプトーム、プロテオーム、メタボロームなどがある。
インシリコ解析:コンピュータを用いたバイオインフォマティクスなどの解析を示す。
オーソログ:種の分岐によって共通の祖先遺伝子から生じた相同な遺伝子のこと。
■ 論文情報
論文タイトル:Phylogenetic distribution and expression pattern analyses identified a divergent basal body assembly protein involved in land plant spermatogenesis
著者:Shizuka Koshimizu, Naoki Minamino, Tomoaki Nishiyama, Emiko Yoro, Mayuko Sato, Mayumi Wakazaki, Kiminori Toyooka, Kazuo Ebine, Keiko Sakakibara, Takashi Ueda and Kentaro Yano
掲載誌:国際雑誌「New Phytologist」
DOI:https://doi.org/10.1111/nph.18385
論文掲載URL:https://nph.onlinelibrary.wiley.com/doi/10.1111/nph.18385
■ 研究者情報(2022年8月現在)
明治大学 農学部
越水静(助教)、矢野健太郎(教授)
基礎生物学研究所 細胞動態研究部門
南野尚紀(特任助教)、海老根一生(助教)、上田貴志(教授)
金沢大学 疾患モデル総合研究センター 研究基盤支援施設
西山智明(助教)
立教大学 理学部生命理学科
養老瑛美子(助教)、榊原恵子(准教授)
理化学研究所 環境資源科学研究センター
佐藤繭子(技師)、若崎眞由美(テクニカルスタッフ)、豊岡公徳(上級技師)
■ 研究支援
本研究はJSPS科研費19H04870(矢野健太郎)、19H04872(海老根一生)、19H05675・21H02515(上田貴志)、20K15824(南野尚紀)、15H04413・19K22448(西山智明)および18K06367(榊原恵子)の支援を受けました。
プレスリリース提供元:@Press